Deutscher Rheumatologiekongress 2025
Deutscher Rheumatologiekongress 2025
Potentiall des nichtüberwachten „Deep-learning“-Verfahrens mit Anwendung der spektralen Clustering-Analyse für Segmentierung der Arthrosonographie-Bilder
Text
Einleitung: Die Segmentierung von Sonographie-Bildern ist ein zentraler Bestandteil der medizinischen Bildverarbeitung, da sie eine präzise Analyse und Diagnose von Geweben und Organen ermöglicht. Die Herausforderungen bei der Sonographie-Bildsegmentierung resultieren aus der variierenden Bildqualität, der Notwendigkeit, unterschiedliche Gewebearten zu unterscheiden, sowie der starken Abhängigkeit von der Bildgeometrie und den Artefakten, die durch das Ultraschallverfahren entstehen [1]. In den letzten Jahren haben fortschrittliche Methoden der künstlichen Intelligenz, insbesondere Convolutional Neural Networks (CNNs), vielversprechende Ergebnisse in der Verbesserung der Segmentierungsgenauigkeit erzielt [2]. In unserer Studie haben wir die nichtüberwachte Segmentierung der Arthrosonographie-Bilder mit spektralem Clustering und Anwendung des Vision Transformers [3] durchgeführt und die Qualität der Segmentierung eingeschätzt.
Methoden: Arthrosonographien wurden mit dem Sonographie-Gerät General Electric LOGIQ E10 durchgeführt. Die Bilder wurden im DICOM-Format gespeichert und exportiert. Anschließend wurden die DICOM-Bilder ins jpeg-Format umgewandelt, sodass die patientenbezogenen und technischen Daten nicht berücksichtigt wurden. Die Bilder der MCP-, PIP- und MTP-Gelenke sowie wurden anschließend kopiert und nach den OMERACT-Kriterien [4] entsprechend der Aktivitätsscore einsortiert. Nur die Bilder im B-Mode ohne Anwendung des Dopplersignals wurden analysiert. Die Sonographie-Bilder mit unzureichender Qualität wurden ausgeschlossen. Um die gleiche Quantität der Bilder pro Gruppe zu erreichen, wurde die Anzahl der Bilder pro Gruppe an die kleinste Gruppe angepasst. Daraufhin haben wir ein Deep-learning Verfahren mit Vision Transformer und spektraler Se[Tab. 1]gmentierung der Bilder angewendet. Nach der Bildersegmentation erfolgte die Einschätzung der Qualität des Verfahrens mit Analyse der Richtigkeit der Segmentierung und deren Homogenität bezüglich der Knochenstruktur, der Grenzen zwischen beiden Knochen eines Gelenkes voneinander, der Grenzen zwischen den Knochen und den Weichteilen, Erkennung der Gelenkkapsel und der Synovialitis. Die Qualität der Segmentierung wurde semiquantitativ entweder mit 0 (wenig Übereinstimmung) 0,5 (maßige Übereinstimmung) oder mit 1,0 (höhere Übereinstimmung) pro Merkmal in jedem Bild eingeschätzt, somit lag die maximale Qualität pro Bild bei 5 Punkten.
Tabelle 1: Ergebnisse der nichtüberwachten Bildersegmentierung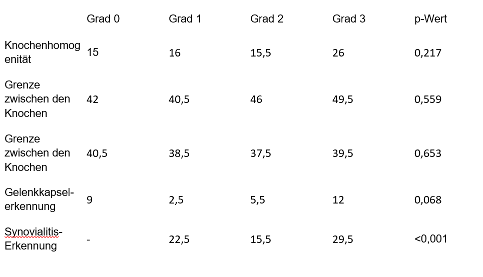
Ergebnisse: 9.897 Sonographie-Bilder wurden aus dem Sono-Gerät extrahiert. 4.660 Bilder im B-Modus ohne Doppleraktivität wurden gespeichert. 1.504 Sonographie-Bilder mit MCP-, PIP- und MTP-Gelenken wurden für die weitere Analyse eingeschlossen, davon: 912 Bilder ohne Hinweise auf Synovialitis, 419 Bilder mit Synovialitis Grad 1, 112 Bilder mit Synovialitis Grad 2 und 61 Bilder mit Synovialitis Grad 3. Die Sonographie-Bilder mit Doppleraktivitätsbestimmung wurden aus der Studie ausgeschlossen. Mittels Randomisierung und Löschen der Extra-Bilder wurde die Anzahl der Bilder pro Gruppe auf 61 reduziert. Das Modell für die Segmentierung wurde anhand von 244 Bildern (61 Bilder pro Gruppe, insgesamt 4 Gruppen) trainiert. Die Segmentierung erfolgte mittels sechs Clustern pro Bild (Abbildung 1 [Fig. 1]). Die Bilder in jeder Gruppe wurden für die Qualität der Segmentierung analysiert, mit maximalem Wert von 61 Punkten pro Merkmal (Tabelle 1 [Tab. 1]).
Abbildung 1: Beispiel der Anwendung des nichtüberwachten „deep-learning“ Verfahrens für Bildsegementierung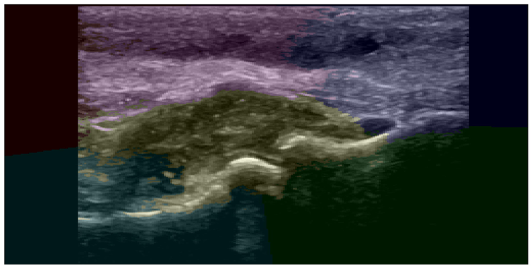
Die Abgrenzung zwischen den Knochen selber sowie zwischen den Knochen und den Weichteilen funktioniert relativ gut bei allen Synovialitis-Graden. Die Homogenität der Knochenstruktur konnte mit dem aktuellen Modell nur mit mäßigem Erfolg erkannt werden und zeigte keinen signifikanten Unterschied zwischen den Gruppen. Die Segmentierung der Synovialitis fiel signifikant unterschiedlich zugunsten der Synovialitis des dritten Grades aus. Die Gelenkkapsererkennung konnte nur in Einzelfällen erfolgen.
Schlussfolgerung: Die Segmentierung der Sonographie-Bilder mittels nichtüberwachter „deep-learning“ spektraler Clustering-Analyse kann einen gewissen Erfolg für Nachweis der Grenzen zwischen den Knochen eines Gelenkes, sowie zwischen den Knochen und Weichteilen und Abgrenzung der Synovialitis verzeichnen.
References
[1] Umbaugh SE. Digital image processing and analysis: computer vision and image analysis. CRC Press; 2023.[2] Kayalibay B, Jensen G, van der Smagt P. CNN-based segmentation of medical imaging data. 2017. arXiv preprint. DOI: 10.48550/arXiv.1701.03056
[3] Tmenova O, et al. Deep Spectral Methods for Unsupervised Ultrasound Image Interpretation.International Conference on Medical Image Computing and Computer-Assisted Intervention. Cham: Springer Nature Switzerland; 2024.
[4] D'Agostino MA, Terslev L, Aegerter P, Backhaus M, Balint P, Bruyn GA, Filippucci E, Grassi W, Iagnocco A, Jousse-Joulin S, Kane D, Naredo E, Schmidt W, Szkudlarek M, Conaghan PG, Wakefield RJ. Scoring ultrasound synovitis in rheumatoid arthritis: a EULAR-OMERACT ultrasound taskforce-Part 1: definition and development of a standardised, consensus-based scoring system. RMD Open. 2017 Jul 11;3(1):e000428. DOI: 10.1136/rmdopen-2016-000428




