70. Jahrestagung der Deutschen Gesellschaft für Medizinische Informatik, Biometrie und Epidemiologie e.V.
70. Jahrestagung der Deutschen Gesellschaft für Medizinische Informatik, Biometrie und Epidemiologie e.V.
Identifizierung von Sepsis-Subtypen mittels maschinellen Lernens auf Basis des Plasma-Proteoms
Text
Einleitung: Jährlich treten in Deutschland rund 280.000 Fälle von Sepsis auf [1], einem lebensbedrohlichen, infektionsgetriebenen systemischen Multiorganversagen. Die Behandlung ist meist auf Antiinfektiva und unterstützende medizinische Maßnahmen limitiert. Spezifische Therapieoptionen, die individuelle molekulare Stoffwechselveränderungen adressieren, stehen aktuell nicht zur Verfügung. Bisher scheiterten molekular-mechanistische Ansätze in klinischen Studien, möglicherweise aufgrund der Komplexität des Syndroms und der heterogenen Kohorten [2], [3]. Mittlerweile ermöglichen Hochdurchsatztechnologien die molekulare Charakterisierung von Patienten. Die DigiSep-Studie z.B. untersucht, ob Diagnostik und Therapie durch Next-Generation Sequencing präzisiert und optimiert werden können [4]. Um die molekulare Grundlage für eine zukünftige personalisierte Behandlung von Patienten mit Sepsis zu schaffen, haben wir eine multizentrische Patientenkohorte mittels Plasma-Proteomics analysiert und molekulare Subtypen in dieser Kohorte mittels maschinellem Lernen identifiziert.
Methoden: Im Rahmen der Studien SepsisDataNet.NRW und CovidDataNet.NRW wurde eine umfangreiche Biobank mit Proben und klinischen Daten kritisch kranker Sepsis-Patienten aufgebaut. Für 333 Patienten wurden Plasmaproben zu zwei Zeitpunkten in der frühen Sepsisphase (Tag 1 und 4) mittels Flüssigchromatographie-gekoppelter Massenspektrometrie analysiert. Eine Hauptkomponentenanalyse (PCA) und anschließendes k-Means-Clustering ermöglichten die Identifikation molekularer Subtypen. Zeitliche Verläufe der Patienten wurde analysiert, indem die Daten des zweiten in den ermittelten Raum der PCA des ersten Zeitpunkts projiziert und den Clustern basierend auf den Abständen zu den Clustermittelpunkten zugeordnet wurden. Ein Random-Forest-Modell mit geschachtelter Monte Carlo Kreuzvalidierung und SHAP-Werte (Shapley Additive Explanations) diente zur Identifikation relevanter Proteine und klinischer Parameter für die Subtypen-Vorhersage. Der Datensatz wurde hierfür stratifiziert in 80/20 Trainings- und Testdaten und das Trainingsset erneut stratifiziert im Verhältnis 80/20 zur Validierung aufgeteilt. Die Performance des Modells wurde mittels Genauigkeit, AUC, Sensitivität, Matthews Korrelation Koefficient, F1-Maß und Präzision bewertet.
Ergebnisse: Es wurden vier Sepsis-Subtypen und deren zeitlicher Verlauf an Tag 1 und 4 der Sepsis identifiziert (Abbildung 1a [Abb. 1]). Die Subtypen unterschieden sich in der Schwere der Erkrankung wobei Cluster 0 die höchste Mortalität (100%) und die höchsten SOFA-Scores aufwies. Cluster 1 war durch eine umfassende Immunreaktion gekennzeichnet, was durch hohe Immunglobulin-Spiegel angezeigt wurde. Die Überschneidung mit unseren Beobachtungen mit [5] deutet auf eine Generalisierbarkeit dieses Subtyps hin. Durch iterative Hinzugabe von Merkmalen in das Modell wurde bestimmt, dass ab 10 Proteine die Steigung der Sensitivität abnimmt (Abbildung 1b [Abb. 1]) und eine begrenzte Anzahl an Proteinen mit hoher prädiktiver Relevanz identifiziert werden (Abbildung 1c [Abb. 1]). Allerdings deutet eine Differenz >0,1 zwischen Trainings- und Testmetriken (z.B. in der Sensitivität 0.167 ± 0.060) auf ein mögliches Overfitting hin.
Abbildung 1: (a) Sankey-Diagramm zur Patientenzuordnung zu Proteom-Subtypen (Tag 1/4) und 30-Tage-Überleben. (b) Streudiagramm: mittlere Sensitivität vs. Anzahl klinischer Merkmale; lila Linie=Protein-only, rote Linie=Knickpunkt. (c) Balkendiagramm: Häufigkeit der Merkmalsauswahl in MCCV mit zehn Proteinen.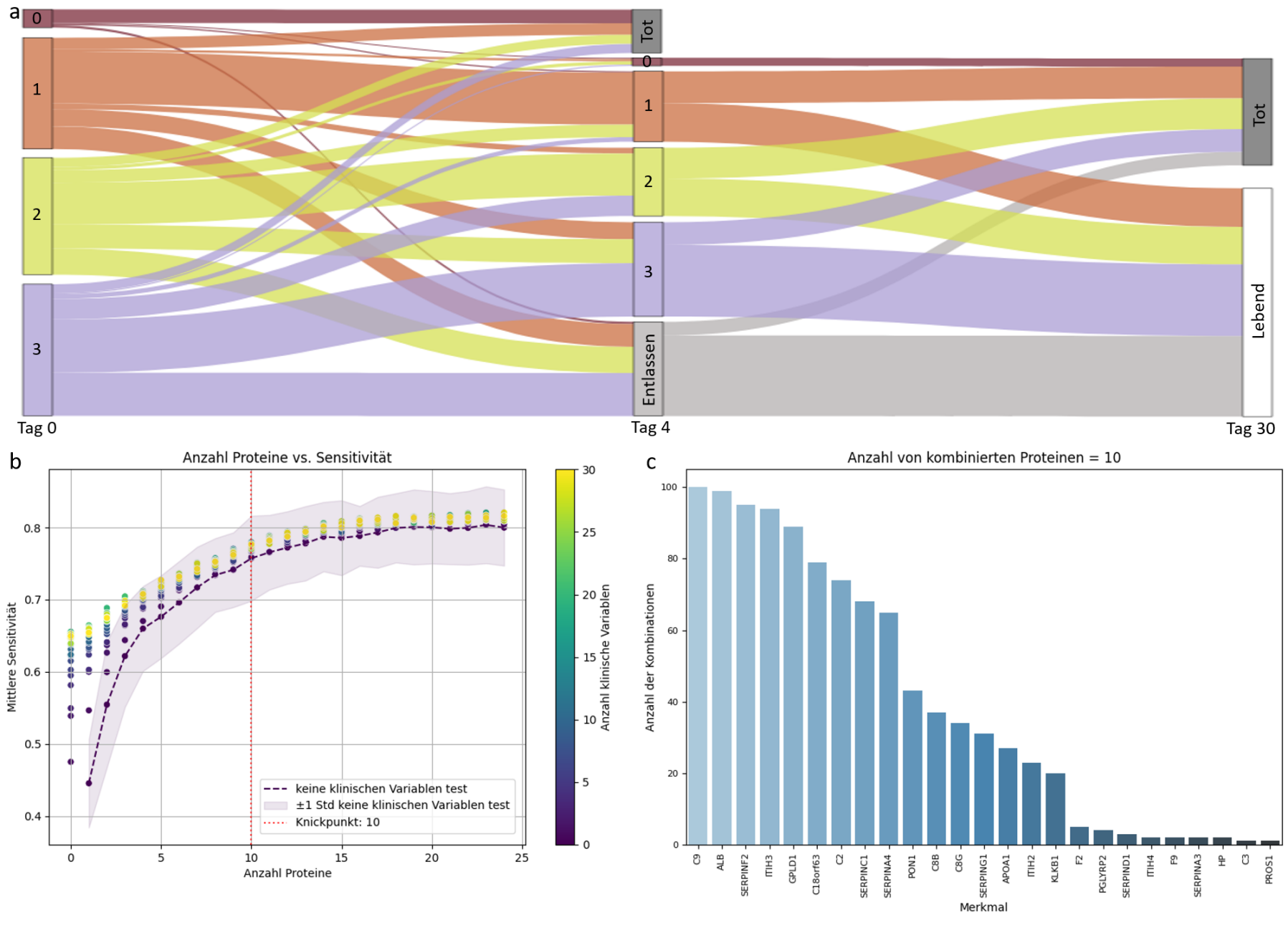
Schlussfolgerung: Die auf Proteom-Ebene identifizierten Subtypen lassen sich nicht allein durch klinische Routineparameter erfassen. Wenige Proteine erlauben jedoch eine Vorhersage der identifizierten Subtypen. Durch das Overfitting des Modells sind die Ergebnisse noch mit Vorsicht zu betrachten und eine weitere Entwicklung des Modells durch eine größere Kohorte bzw. externe Validierung dieser Ergebnisse ist notwendig. Die proteombasierten Unterschiede liefern neue Einblicke in die molekularen Mechanismen der Sepsis und schaffen die Grundlage für eine Integration klinischer und molekularer Patientendaten. Dieser Ansatz kann die Grundlage für die zukünftige Entwicklung personalisierter, zielgerichteter Therapien gegen Sepsis ermöglichen.
Die Autoren geben an, dass kein Interessenkonflikt besteht.
Die Autoren geben an, dass ein positives Ethikvotum vorliegt.
Literatur
[1] Fleischmann-Struzek C, Schwarzkopf D, Reinhart K. Inzidenz der Sepsis in Deutschland und weltweit: Aktueller Wissensstand und Limitationen der Erhebung in Abrechnungsdaten. Med Klin Intensivmed Notfmed. 2022;117(4):264–8. DOI: 10.1007/s00063-021-00777-5[2] Hotchkiss RS, Moldawer LL, Opal SM, Reinhart K, Turnbull IR, Vincent JL. Sepsis and septic shock. Nat Rev Dis Primers. 2016;2(1): 16045. DOI: 10.1038/nrdp.2016.45
[3] Boomer JS, Green JM, Hotchkiss RS. The changing immune system in sepsis: Is individualized immuno-modulatory therapy the answer? Virulence. 2014;5(1):45–56. DOI: 10.4161/viru.26516
[4] Brenner T, Skarabis A, Stevens P, Axnick J, Haug P, Grumaz S, Bruckner T, Luntz S, Witzke O, Pletz MW, Ruprecht TM, Marschall U, Altin S, Greiner W, Berger MM; TIFOnet Critical Care Trials Group. Optimization of sepsis therapy based on patient-specific digital precision diagnostics using next generation sequencing (DigiSep-Trial) — study protocol for a randomized, controlled, interventional, open-label, multicenter trial. Trials. 2021;22:714. DOI: 10.1186/s13063-021-05654-4
[5] Mi Y, Burnham KL, Charles PD, Heilig R, Vendrell I, Whalley J, Torrance HD, Antcliffe DB, May SM, Neville MJ, Berridge G, Geoghegan CG, Radhakrishnan J, Nesvizhskii AI, Yu F, Investigators G, Davenport EE, McKechnie S, Davies R, O’Callaghan DJP, Patel P, del Arroyo AG, Karpe F, Gordon AC, Ackland GL, Hinds CJ, Fischer R, Knight JC High-throughput mass spectrometry maps the sepsis plasma proteome and differences in patient response. Science translational medicine. 2024;16(750):eadh0185. DOI: 10.1126/scitranslmed.adh0185




